[분자 생물학] Cloning - restriction enzyme
안녕하세요~ 이번 포스팅은 cloning입니다.
저는 처음에 cloning에 대한 첫인상은 굉장히 오래걸리고 어렵다였습니다.
그래서 그런지 뭔가 처음 시도하는 것 자체가 굉장히 어려웠습니다.
하지만 생각보다 어렵진 않아요!! (잘 안될 뿐!)
과정이 어렵진 않다는 겁니다..ㅎㅎ
cloning 방법도 굉장히 여러 가지입니다.
우선 이번에는 restriction enzyme을 이용한 cloning에 대해 설명드릴게요!!
cloning을 하는 이유는 굉장히 많죠.
overexpression, viral vector를 이용한 shRNA 발현, cas9과 guide RNA 발현 등 너무나 많습니다.
이번 포스팅은 정확하게 말하자면 overexpression을 위한 cloning입니다.
제가 있던 실험실에서는 overexpression을 위한 vector들이 많았습니다.
대표적으로 CMV promoter가 있는 pCMV류나 pcDNA 종류죠.
이들은 promoter들이 굉장히 강력해서 down stream에 있는 gene 발현이 굉장히 셉니다.
pCMV 공벡터에 EGFR cDNA를 넣는 다고 가정하고 예시로 설명드리겠습니다.
우선 plasmid에 발현시키기 위해선 gene 전체를 옮기는게 아닙니다. exon 부분만을 옮기게 되는데
gene 전체를 옮기는 것은 사이즈가 너무 커서 pCMV같은 plasmid에 옮기는 것 자체가 불가능합니다.
gene 전체를 옮기려면 BAC 같은 capacity가 큰 vector에 옮겨야합니다.
exon 부분만을 옮기려면 어떻게 해야할까요??
바로 cDNA(complementary DNA) library를 이용하는 것입니다.
mRNA의 양끝에는 5'-UTR, 3'-UTR이 있고 그 사이에 splicing되어 exon들만 남아있게 됩니다.
cDNA는 mRNA를 주형으로 dNTP를 이용하여 만들어집니다.
우리는 cDNA를 이용해서 exon부분만을 타겟하여 primer를 제작하고 PCR을 통해 exon 부위만을 증폭시켜 cloning을 진행하면됩니다.
cDNA를 만드는 과정은 나중에 포스팅하기로 하겠습니다.
cDNA까지 얻었다는 가정 하에 우리가 원하는 gene을 얻기 위해 primer를 제작해야합니다.
일반적으로 gene의 정보는 ncbi에 접속해서 얻으면 되는데요
(https://www.ncbi.nlm.nih.gov/gene/) 여기로 접속하시면 됩니다.

gene을 검색하시면 gene에 대한 정보가 나옵니다.

검색하면 아래와 같이 Search results가 나오는데 우리는 human의 gene이 필요하니까 맨 위를 누르시면 됩니다.

눌렀더니 정보가 엄청나게 많이 나옵니다;;
위와 같이 NCBI Reference Sequences (RefSeq)을 가보면 EGFR에 관한 mRNA와 protein 정보들이 나오는데 저렇게 많은 이유는 splicing으로 인한 transcript variant들이 여러 개 존재하기 때문입니다.
transcript variant에 따라 간혹 function이 다르거나 function의 유무가 다르기도 합니다.
어지간하면 full-length가 거의 우세한 transcript variant이고 대부분 full length가 모든 functional한 domain, motif를 가지고 있기 때문에 우리는 full-length를 cloning 해보도록 합시다.
transcript variant를 정했으면 primer design을 해야하는데요,
EGFR full-length의 CDS 정보를 copy해서 snapgene이라는 파일에 옮겨봅니다.

CDS의 정보는 CCDS (Consensus CDS)에 들어가면 찾을 수 있고 방금 전 gene 검색 후 나온 창에서 내려가다보면 우측에 CCDS라고 써있는 부분을 클릭하시면 CCDS를 볼 수 있는 화면으로 넘어갑니다.

CCDS로 넘어오면 여러가지 버전의 CCDS들이 있습니다.
어떤 걸 선택해도 상관없으므로 전 맨 아래 CCDS를 선택했고 위에 보시면 Links에서 C를 누르시면 CDS 정보가 나옵니다.

위 그림과 같이 EGFR의 DNA sequence 정보를 찾을 수 있고 전체 sequence를 copy하고 snap gene으로 paste합니다.
snapgene은 써보신 분들은 알겠지만 DNA work할 때 굉장히 편리한 프로그램입니다.

Snap gene을 열고 New file 클릭 후 EGFR CDS를 paste해줍니다.
primer를 짜기 위해 5'-end와 3'-end에서 각각 forward와 reverse를 짜줍니다.
대략 24bp정도씩 우선 짜보면


Forward - 5'-ATGTTCAATAACTGTGAGGTGGTC-3'
Reverse - 5'-TCATGCTCCAATAAATTCACTGCT-3'
가 됩니다.
이대로 primer를 디자인 하고 PCR을 돌리면 될까요??
아닙니다!!
cDNA library에는 수많은 gene들이 존재하므로 EGFR외에 다른 gene들이 primer에 의해 amplification될 수 있습니다.
오로지 EGFR에만 붙는 specific 한 primer를 제작하기 위해 primer blast라는 과정을 거칩니다.
primer blast는 내가 디자인한 primer로 어떤 CDS가 나올지 찾아주는 프로그램이고 역시 NCBI에서 제공합니다. (갓NCBI !!)
구글에서 primer blast라고 검색하시거나 여기로 들어가시면 됩니다. (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)

디자인한 primer를 위와 같이 5'->3' 순서로 primer parameters에 넣어줍니다.
그리고 아래 쭉 내려가면

Get primers 를 클릭해줍니다.
대략 몇 분 단위로 걸리는데 1분 내외인 것 같습니다.

운이 좋게도 CDS 양 끝의 24bp씩 따서 primer 디자인 했는데 amplicon 결과들이 EGFR만 뜨네요!! (오늘 로또 각)
EGFR의 transcript variant가 여러개 나오는 이유는 exon 1번과 끝 exon이 존재하는 EGFR transcript variant가 여러개 이기 때문입니다.
PCR을 해보면 내가 얻은 cDNA library에서 transcript variant가 어떤 것인지는 모르겠지만 우세한 variant가 PCR에서 amplification될 확률이 큽니다. (어지간해선 여러 개 나올 일도 없긴합니다만)
여기서 추가적으로 우린 restriction enzyme을 사용하기 위해 primer의 끝에 restriction enzyme이 인식하는 site를 추가적으로 붙여줘야합니다.
일반적으로 plasmid의 Multi Cloning Site(MCS)에서는 굉장히 많은 restriction enzyme site가 있습니다.
주로 많이 쓰는 restriction enzyme은 EcoR1, Hind3, Kpn1, Not1, BamH1 등이 있습니다.
이 중에서 맘에드는 걸로 고르면 됩니다.
단!! CDS에도 restriction enzyme이 있을 수 있으니 CDS에 없는 restriction enzyme을 골라야합니다.
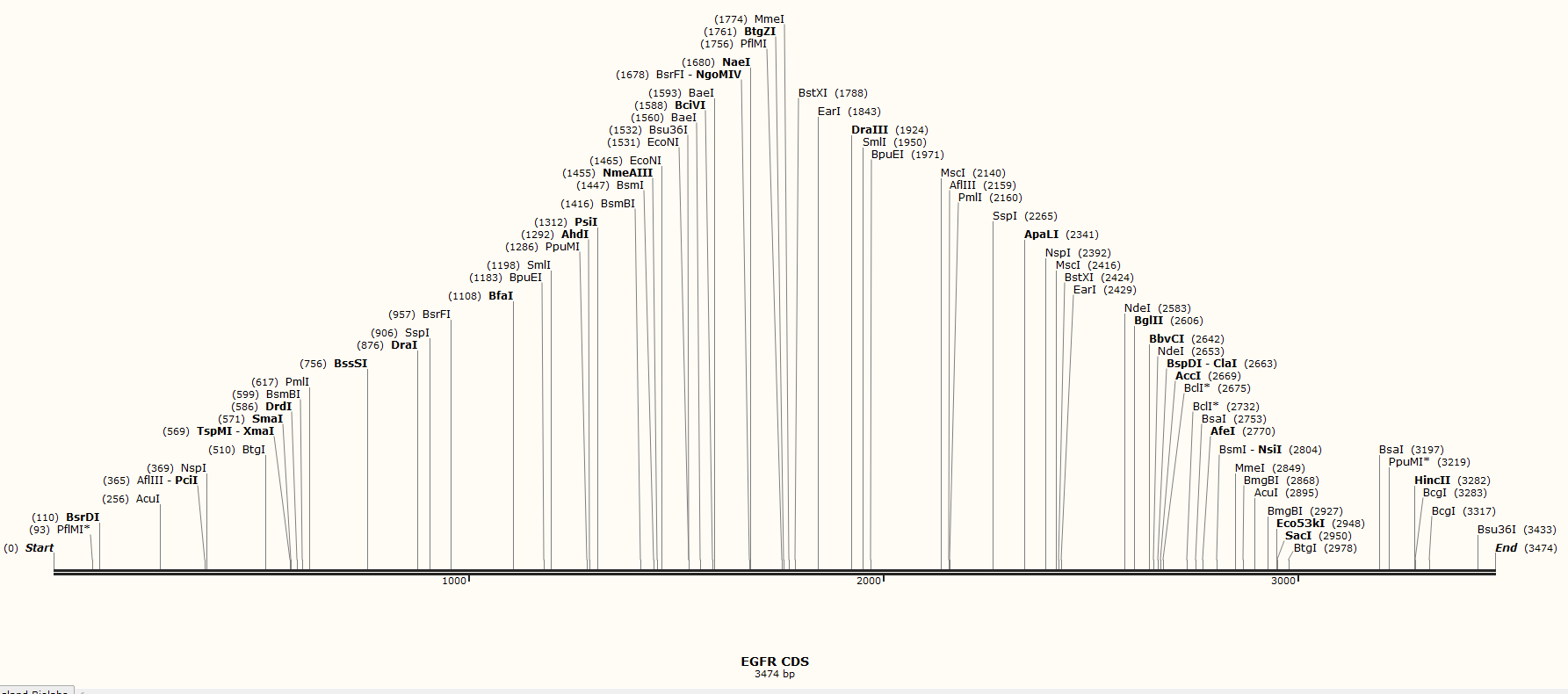
Snapgene을 이용하면 위와 같이 restriction site를 확인할 수 있습니다. 엄청 많죠??
여기서 딱히 걸리는 건 없네요.
그러면 5'-end에는 EcoR1 site를 만들어주고 3'-end에는 BamH1 site를 만들어서 primer를 modify해봅시다.
일반적으로 restriction enzyme은 palindrome sequence(회문구조를 가진 배열)이기 때문에 5',3' 굳이 상관 안하셔도 됩니다.
EcoR1 site는 GAATTC BamH1은 GGATCC입니다.


primer에 5'-end에 restriction enzyme site를 달아주면
Forward - 5'-GAATTCATGTTCAATAACTGTGAGGTGGTC-3'
Reverse - 5'-GGATCCTCATGCTCCAATAAATTCACTGCT-3'
가 됩니다.
자 이렇게 primer 디자인이 끝났습니다~ 라고 하면 섭하죠.
물론 이렇게 해도 되긴합니다만, biology는 우리 생각처럼 되지 않을 때가 있죠.
plasmid같이 enzyme site 양옆으로 DNA sequence가 쭉 있다면 상관 없지만
우린 PCR로 EGFR gene을 본뜬 뒤 양 끝을 EcoR1과 BamH1으로 잘라줘야합니다.
PCR amplicon은 double strand DNA이고 linear합니다.
즉, 양 끝이 있다는 점이죠.
restriction enzyme은 양끝에 만 떡하니 restriction enzyme site가 있으면 효율이 떨어집니다. 많이많이
그래서 plasmid 처럼 양끝에 sequence를 조금 더 달아줘야합니다.
restriction enzyme cut의 efficiency를 올리기 위해 primer 양 끝에 sequence를 조금 더 추가해야한다는 점이죠.
아무 sequence나 달아주는 것은 아니고 NEB에서 제공하는 Cleavage Close to the End of DNA Fragments (oligonucleotides) 를 참고해야합니다.
찾기 쉽게 링크 복붙해 봅니다..!!
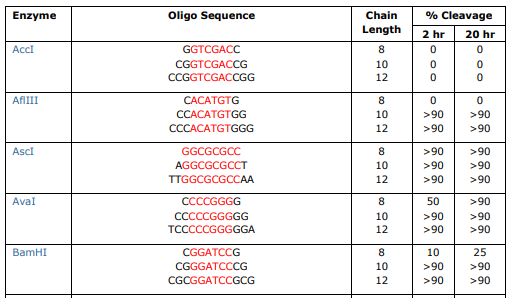
자료를 보면 BamH1이 efficiency의 90%이상을 보이려면 GGATCC sequence 양 옆으로 CG가 붙어야 합니다.EcoR1도 똑같이 GAATTC 양 옆으로 최소 CG가 붙어야 cut efficiency가 90%이상이 됩니다.
그래서 우리의 primer 최종 design은
Forward - 5'-CGGAATTCCGATGTTCAATAACTGTGAGGTGGTC-3' (EcoR1)
Reverse - 5'-CGGGATCCCGTCATGCTCCAATAAATTCACTGCT-3' (BamH1)
가 되겠습니다.
primer를 주문하고 cDNA를 주형으로 PCR을 시작합니다.
PCR protocol은 제가 포스팅한 것을 참고하셔도 좋고 다른 좋은 사이트들이 많이 있으니 참고해서 PCR을 진행하시면 될 것 같습니다.
PCR 후 1.5% Agarose gel 을 이용해서 PCR이 제대로 떠졌으면 EGFR CDS size인 3.4~3.5 kb 인근에서 band가 보일 것입니다.
여기서 어지간하면 1 band로 뜨고 여러 밴드가 뜰 수도 있습니다.
원인은 여러가지겠지만 PCR condition이 안잡혔을 확률이 큽니다.
이미 primer blast를 진행해서 EGFR만 나오는 것을 확인했기 때문에 다른 gene이 amplification될 일은 거의 없습니다.
1 band인 것을 확인하면 PCR amplicon과 사용할 vector pCMV를 EcoR1, BamH1을 이용해서 잘라줍니다.
restriction enzyme을 이용한 cut은 조만간 포스팅 하도록 하겠습니다.
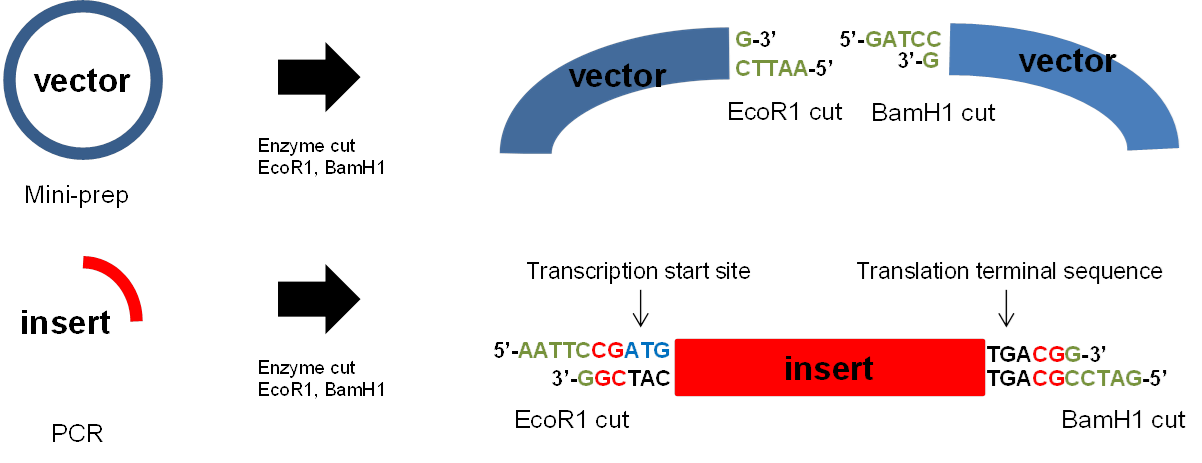
enzyme cut을 하고 나면 잘린 PCR amplicon과 잘린 vector를 얻어야하기 때문에 Agarose gel에 내린 후 gel extraction을 진행해줍니다.
gel extraction 또한 제가 나중에 포스팅을... ㅠㅠ
gel extraction하여 얻어진 친구들의 농도를 측정해준 뒤 ligation 해줍니다.

ligation의 경우 vector 와 insert의 비를 1:2~1:3 정도로 잡아주면 됩니다. ( 농도 비)
ligation 후 DH5a에 transformation하여 cloning 여부를 확인합니다.
cloning 과정이 사실 굉장히 복잡한데 조금 스킵한 곳도 좀 있는 것 같습니다.
포스팅 후 다른 protocol들도 정리하면서 조금씩 정리하고 자세한 protocol도 업로드 하겠습니다.
글이 너무 길어져서 나중에 다시 편집해야겠네요..
cloning 꼭 모두들 성공합시다~!